Advances in Microbiology
Vol.3 No.02(2014), Article ID:13691,11 pages
DOI:10.12677/AMB.2014.32006
Cloning and Sequence Analysis of Laccase Gene from Pleurotus nebrodensis
Key Laboratory of Ion Beam Bioengineering of Autonomous Region, Inner Mongolia University, Hohhot
Email: *guojf101@sina.com
Copyright © 2014 by authors and Hans Publishers Inc.
This work is licensed under the Creative Commons Attribution International License (CC BY).
http://creativecommons.org/licenses/by/4.0/



Received: Apr. 28th, 2014; revised: May 29th, 2014; accepted: Jun. 5th, 2014
ABSTRACT
Pleurotus nebrodensis as a rare mushroom, it is necessary to improve its yield and quality; laccase has an important influence on the growth of fungi. A pair of specific primers was designed by conservative sequence of pleurotaceae laccase gene, and the laccase cDNA sequence of Pleurotus nebrodensis was amplified by RT-PCR. In order to obtain the laccase gene, taking genomic DNA as test material, 2606-bp-long laccase gene fragment was cloned by PCR. Through purification, the DNA is cloned to the pGM-T; after screening, PCR identification and the sequence analysis, it is proved that the fragment is complete laccase DNA (GenBank KC789845). Through the intron and exon analysis, the analysis results consistent with the cDNA sequence obtained from the reverse transcription of sequencing. It contains one open reading frame (ORF), which encodes for a polypeptide containing 519 amino acids (Genbank AGO64758.1). The result of protein blast showed that the gene had three Cu-oxidase conserved domains, and the highly conserved laccase signature sequence L3 was also found from the gene sequence. The secondary structure and three-dimensional structure of the enzyme were predicted by bioinformatics software.
Keywords:Pleurotus nebrodensis, Laccase Gene, Cloning, Sequence Analysis

白灵菇漆酶基因的克隆及其序列分析
薛 丹,刘 腾,那 日,王志永,郭九峰*
内蒙古大学自治区离子束生物工程重点实验室,呼和浩特
Email: *guojf101@sina.com
收稿日期:2014年4月28日;修回日期:2014年5月29日;录用日期:2014年6月5日

摘 要
白灵菇作为一种珍稀食用菌,提高其产量及质量具有重要的现实意义,而漆酶对真菌的生长发育具有重要影响。本文根据侧耳科漆酶基因共同保守序列,设计引物,利用RT-PCR的方法,克隆白灵菇漆酶序列,经测序获得其cDNA序列。为进一步获得该漆酶基因序列,以白灵菇基因组DNA为模板,PCR扩增出长2606 bp的漆酶基因DNA片段,DNA经纯化后克隆到pGM-T载体上,经筛选、PCR鉴定、序列分析,证明该片段为完整的白灵菇漆酶基因(GenBank注册号KC789845)。对基因序列的内含子/外显子进行分析,分析结果与经反转录测序获得的cDNA序列一致。该基因的开放阅读框由1596个核苷酸组成,编码一个由531个氨基酸组成的多肽(GenBank登录号AGO64758.1),Protein Blast 分析结果表明,该基因含有3个铜氧化酶(Cu-oxidase)保守结构域,并具有高度保守的漆酶特征序列L3;又进一步对白灵菇漆酶二级结构进行分析,并通过 SWISS-MODE预测了该酶的三维结构。
关键词
白灵菇,漆酶基因,克隆,序列分析

1. 引言
食用菌是可供人类食用的大型真菌,是无公害的绿色健康食品,白灵菇(Pleurotus nebrodensis),学名白灵侧耳,隶属于真菌门,担子菌亚门(Basidiomycolina)真菌纲(Hymenornycetes)伞菌目(Agaricales)侧耳科(Pleurotaceae)侧耳属(Pleurotus),是近年来逐渐发展起来的食用菌新秀之一[1] ,是食用和药用价值都很高的珍稀食用菌,但其产量和质量都有待进一步提高。漆酶对食用菌生长具有重要作用,可降解培养基中的木质素,为菌体提供营养以使其生长发育,漆酶也参与菌筒转色,因此影响着成品的产量及质量[2] ,同时漆酶在菇类菌体生长发育的过程中起到抑制杂菌污染的作用[3] ,从而加快了菌丝生长速度,缩短了制种时间,提高了食用菌的产量及质量。漆酶(Laccase.EC.1.10.3.2)是一种含铜多酚氧化酶[4] ,多分布于植物[5] 和真菌[6] 中。其中,白腐真菌生产的漆酶被认为效果最好。漆酶在木质素的生物降解、造纸工业、食品加工、废水处理、土壤净化、染料脱色、生物传感器、免疫检测等方面具有广泛的应用价值[7] -[11] 。伴随着分子生物学技术的发展,已有许多漆酶基因被克隆和测序[12] ,一些漆酶基因也实现了异源表达。但迄今尚未见有关白灵菇漆酶基因的研究报道,本试验通过对白灵菇漆酶基因的克隆,系统分析该基因序列,旨在为进一步研究漆酶酶学性质及该基因的生物学功能和工程菌的构建奠定理论基础。
2. 材料与方法
2.1. 菌株和质粒
白灵菇(Pleurotus nebrodensis)由内蒙古农牧业科学院菌种保藏中心提供,大肠杆菌感受态细胞TOP10,pGM-T载体等由内蒙古大学离子束生物实验室保存。
2.2. 培养基及缓冲液的配制
真菌培养基配方:蛋白胨5 g,酵母浸出粉2 g,蔗糖30 g,磷酸二氢钾1 g,硫酸镁0.5 g,蒸馏水1000 mL,固体培养基加入0.8%的琼脂,pH = 6.0。灭菌条件:121℃、25 min。
RB亮蓝真菌培养基:配置RB亮蓝母液(5.0 mg/mL),取10 mL加入到90 mL含有100 mL的真菌培养基的药品的培养基中。
大肠杆菌(E.coli)培养:LB液体、固体培养基。
0.1 mol/L NaAC母液的配制:称取醋酸钠0.41 g,用蒸馏水定溶50 mL。0.1 mol/L醋酸母液的配制:吸取冰醋酸5.72 mL,用蒸馏水定溶1000 mL。将两种母液混合为浓度0.1 mol/L HAc-NaAc缓冲液(pH 4.0)。
0.5 mmol/LABTS溶液的配制:取0.137 g的ABTS溶解于500 mL的HAc-NaAc缓冲液中。
2.3. 试剂
Agar购于BIOSHARP公司,Agar Rose购于SHANGHAI YITO BIO-INSTRUMENT公司,X-gal、IPTG、AMP购于TaKaLa公司,RB亮蓝、ABTS购于sigma公司,核酸染料购于Bio Teke公司,其余试剂为国产分析纯级。
LongRang 2Step RT-PCR Kit购于QIAGEN公司,2Taq PCR Master Mix,DNA Marker D2000,pGM-T克隆试剂盒,普通琼脂糖凝胶DNA回收试剂盒(离心柱型),植物基因组DNA提取试剂盒(离心柱型),植物总RNA提取试剂盒(离心柱型)均购于TIANGEN BIOTECHBEIJING)CO.LTD,PCR引物由上海生工生物工程公司合成。
2.4. 漆酶活性测定
2.4.1. 定性测定
选取多种食用菌的菌株接种于RB亮蓝培养基中就行筛选,20℃进行培养,菌丝生长良好,使RB亮蓝变色能力强、变色速度快的平板即为酶活性较高的菌株。
2.4.2. 定量测定
漆酶活性测定参照文献[13] 采用ABTS法,并加以改动。配制0.5 mmol/LABTS溶液,将ABTS用0.1 mol/L HAc-NaAc缓冲液(pH 4.0)溶解。5.0
mL反应体系,由于酶活高低不同所加入的酶液量也不同,多次实验选取最佳量。在室温(25℃)下反应测定3 min内反应液在420 nm处吸光度的增加值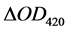 。定义该条件下,每分钟催化1 μmol
ABTS氧化所需酶量为1个酶活力单位(U),并按下式计算漆酶活力:
。定义该条件下,每分钟催化1 μmol
ABTS氧化所需酶量为1个酶活力单位(U),并按下式计算漆酶活力:

其中,N为稀释倍数; 为3 min内反应液在420 nm处吸光度的增加值;36,000为ABTS氧化态的摩尔吸光系数(L·mol−1·cm−1)。
为3 min内反应液在420 nm处吸光度的增加值;36,000为ABTS氧化态的摩尔吸光系数(L·mol−1·cm−1)。
2.5. PCR引物的设计
根据伞菌目侧耳科漆酶基因共同保守序列,设计引物P和Q(包含起始密码子和终止密码子),并且引入BglⅡ和BstEⅡ酶切位点,以总RNA的反转录cDNA为模板克隆漆酶基因的完整ORF的cDNA序列。同时,为了测量漆酶基因DNA序列,根据真菌漆酶保守序列并考虑到基因片段过长的因素,设计两对特异性引物F、G1、C、R,以白灵菇基因组DNA为模板克隆获得白灵菇漆酶基因DNA序列,并进行测序。设计引物序列如表1所示。
2.6. PCR扩增
2.6.1. RT-PCR获得漆酶基因cDNA序列
提取白灵菇总RNA模板,用反转录试剂盒(LongRang 2Step RT-PCR Kit,QIAGEN)进行RNA的反
表1. 克隆白灵菇菌株漆酶基因DNA序列和cDNA序列所用引物
转录,合成第一链cDNA。克隆漆酶基因,以cDNA第一条链为模板,反应体系为:10x LongRange PCR Buffer 5 μL,dNTP mix(10 mM each)2.5 μL,Primer A(10 μM)1 μL,Primer B(10 μM)1 μL,Rnase-free water 39.1 μL,LongRang PCR Enzyme Mix 0.4 μL,cDNA模板1 μL,共50 μL体系。反应条件为:93℃ 3 min;93℃ 15 s,62℃ 30 s,68℃ 1.5 min,35个循环;4℃保存。扩增产物用1.0%的琼脂糖凝胶电泳鉴定。
2.6.2. PCR扩增漆酶基因DNA片段
以白灵菇基因组DNA为模板进行扩增,PCR反应体系如下:模板DNA1.5 μL,2Taq PCR Master Mix 10 μL,上游引物(10 pmol/μL)1 μL,下游引物(10 pmol/μL)1 μL,无菌水6.5 μL,总体积20 μL。PCR反应条件为:95℃ 5 min;95℃ 15 s,F-C:67℃ 30 s/G1-R:63℃ 30 s,72℃ 1.5 min,35个循环;72℃ 7 min。用1.0%的琼脂糖凝胶电泳鉴定。
2.7. PCR产物回收、克隆及鉴定
PCR产物经1.0%琼脂糖凝胶电泳后,切下目的条带,用普通琼脂糖凝胶DNA回收试剂盒进行回收。利用pGM-T克隆试剂盒,将回收产物与pGM-T载体进行连接,连接产物转化到感受态细胞大肠杆菌中,然后涂在含Amp、IPTG、X-gal的LB琼脂平板上,37℃培养过夜。随机挑取白色菌落进行PCR方法检测插入片段是否正确。
2.8. 测序及序列分析
DNA测序由上海生工生物公司完成,以T7、SP6为测序引物进行双向测序,每个单菌落进行三次重复试验。序列分析使用DNAMAN、NcBI网站上(http://www.ncbi.nlm.nih.gov)BLAST软件及内含子/外显子分析预测软Augustus(http://bioinf.uni-greifswald.de/augustus/),采用英国瓦卫克大学PSIpred服务器(http://bioinf.cs.ucl.ac.uk/psipred/)对漆酶的蛋白质序列进行二级结构在线分析,同时递交至
(http://swissmodel.expasy.org/docs/structures_models)中进行漆酶氨基酸序列三维结构的同源建模,对漆酶蛋白信号肽预测使用工具Signal
(http://www.cbs.dtu.dk/services/SignalP/),使用网络DAS服务器
(http://www.sbc.su.se/~miklos/DAS)对漆酶蛋白序列进行跨膜结构预测。
3. 结果与分析
3.1. 漆酶高产菌株的筛选及漆酶活性测定
根据RB亮蓝的变色能力,从多种食用菌株挑选三种菌株,使RB亮蓝变色能力强、中、弱的菌株,为进一步验证,采用ABTS法定量测定三种菌株的漆酶活性,测定结果显示,与定性测量结果吻合,证实白灵菇漆酶的酶活性最高,酶活性为143.98 U/L,为进一步克隆白灵菇漆酶基因提供依据。
3.2. 总RNA的提取及漆酶基因cDNA 序列的克隆
提取白灵菇菌丝体总RNA进行了1%琼脂糖电泳检测结果,如图1所示。28S和18S的两条亮带都比较清晰,并且28S的亮度大约是18S的两倍,说明提取的总RNA完整性较好。同时用超微量分光光度计测量OD260/OD280读数为2.046,说明提取的RNA质量较高,适用于后续的RT-PCR实验。
以提取的总RNA为模板,利用LongRang 2Step RT-PCR Kit合成cDNA第一链,利用引物P和Q,以反转录合成的第一链cDNA为模板,克隆白灵菇漆酶基因cDNA序列,经PCR扩增得到1600 bp左右的条带,回收PCR产物,将其与pGM-T载体连接、转化感受态细胞、阳性克隆筛选,随机挑取3个阳性克隆进行PCR鉴定,从图2可以看出,利用内外引物进行PCR扩增均可得到相应长度的片段,以外引物(T7、SP6)进行测序,得知该片段是由1765 bp组成的开放阅读框全长序列。分析得出该基因的cDNA序列完整开放阅读框的位置在76~1672 bp,起始密码子为ATG,终止密码子为TAG。
3.3. 漆酶基因DNA片段的克隆及序列分析
以白灵菇基因组DNA作为模板,分别扩增基因序列的两段。从图3可以看出第一段基因长度大约都为1800 bp左右,第二段基因长度大约为1200 bp左右,分别回收PCR产物,将其与pGM-T载体连接、转化感受态细胞、阳性克隆筛选,随机挑取3个阳性克隆进行PCR鉴定,得到插入片段序列结果一致的
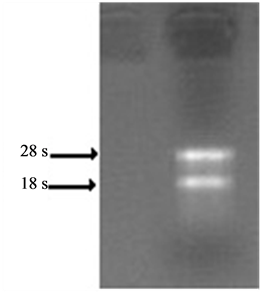
Figure 1. Agarose gel analysis of total RNA
图1. 白灵菇总RNA琼脂糖凝胶电泳图

Figure 2. Amplification cDNA fragments of Laccase gene
图2. 漆酶基因cDNA片段的扩增

Figure 3. The PCR amplification of laccase gene from Pleurotus nebrodensis
图3. 白灵菇漆酶基因DNA片段的PCR扩增
克隆。说明漆酶基因片段已成功克隆进载体pGM-T中,同时送上海生工生物工程公司进行测序,得到白灵菇漆酶基因的两段序列。利用DNAMAN进行序列拼接得到白灵菇漆酶基因组序列,将序列提交至Genebank,登录号:KC789845。
通过内含子/外显子分析预测软件Augustus对漆酶基因DNA序列进行分析,其中有19个间隔区,确定为内含子区,推测出漆酶片段的编码区和非编码区,并且外显子和内含子边界符合GU-AG规则,预测出漆酶基因cDNA片段长度为1596 bp,使用DNAMAN与经反转录测序获得的cDNA序列进行比对,发现比对结果完全一致。进一步证实了漆酶基因的开放阅读框由1596个核苷酸组成,起始密码子为ATG,终止密码子为TAG,编码了一个由531个氨基酸组成的多肽,如文后附图所示。
3.4. 氨基酸序列分析及系统进化树的构建
漆酶基因编码了一个由531个氨基酸组成的多肽,推测的分子量大小为56727.1,等电点pI为4.56,有9个可能的N-糖基化位点(Asn-Xaa-Thr/Ser),应用SignalP4.1 Server在线对白灵菇漆酶蛋白进行信号肽预测,均发现在氮端存在1个信号肽(MFPGARILATLTLALHLLHGTHAA),信号肽潜在的剪切位点可能位于第23和第24个氨基酸之间,如文后附图所示。使用网DAS服务器(http://www.Sbc.Su.se/~miklos/DAS)对白灵菇漆酶蛋白序列进行跨膜结构预测[14] ,在strict cut-off 严格条件限制下,白灵菇漆酶蛋白在8~19 氨基酸之间存在1个跨膜区域,如图4示。
通过NCBI(http://blast.ncbi.nlm.nih.gov/Blast.cgi)对漆酶氨基酸序列进行Protein Blast,与NCBI数据库中的氨基酸序列进行同源性比对。Protein Blast 分析结果表明,该漆酶基因含有3个铜氧化酶(Cu-oxidase)保守结构域,分别为Cu-oxidase-3、Cu-oxidase和Cu-oxidase-2,GeneBank登录号(pfam07732、pfam00394、pfam00731),序列分别位于附图中的64~165、177~317、378~504氨基酸残基处,下划线表明,并具有高度保守的漆酶特征序列L3。
应用DNAMAN软件对漆酶氨基酸序列(GeneBank登录号AGO64758.1)与糙皮侧耳(Pleurotus ostreatus)漆酶(GeneBank登录号Q12729.1)、杏鲍菇(Pleurotus eryngii)漆酶(GeneBank登录号ADD14077.1)、紫孢侧耳(Pleurotus sapidus)(GeneBank登录号CAH05069.1)、桃红侧耳(Pleurotus salmoneostramineus) (GeneBank登录号BAI66142.1)、凤尾菇(Pleurotus sajor-caju) (GeneBank登录号CAD45377.1)、铜绿球盖菇 (StropHaria aeruqinose)(GeneBank登录号AFE48786.1)、毛头鬼伞(Coprinus comatus) (GeneBank登录号AFD97050.1)、黑蛋巢菌(Cyathus bulleri) (GeneBank登录号ABW75771.2)、香菇(Lentinula edodes)漆酶(GenBank登录号BAB83131.1)等9个漆酶氨基酸进行序列比对,并构建系统进化树,如图5所示。
3.5. 漆酶二级结构分析及三维结构建模
通过PSIpred服务器(http://bioinf.cs.ucl.ac.uk/psipred/)对漆酶的蛋白质序列进行二级结构在线分析,白灵菇漆酶二级结构主要是由无规则卷曲50.9%;β-折叠43.6%;α-螺旋5.5%构成。通过蛋白质氨基酸序列及二级结构预测分析,与已知蛋白的三维结构进行同源模建,通过SWISS-MODEL预测了白灵菇漆酶的三维模型,如图6所示。

Figure 4. Transmembrane domain of Pleurotus nebrodensis laccase amino acid sequence predicted by DAS server T
图4. DAS 服务器对白灵菇漆酶氨基酸序列跨膜区域的预测

Figure 5. Phylogenetic tree analysis of laccase primary
图5. 漆酶一级结构的系统进化树分析

Figure 6. Three-dimensional structure of Pleurotus nebrodensis laccase predicted by SWISS-MODEL
图6. 通过SWISS-MODEL预测的白灵菇漆酶三维模型
4. 讨论
中国食用菌资源丰富,也是最早栽培、利用食用菌的国家之一,研究发现很多食用菌菌体中富含漆酶,在菌体生长过程中参与木质素及其相似结构的降解,在自然界生物学降解和物质循环中起着重要的作用,同时白腐菌分泌漆酶的能力非常强,是目前获得漆酶的最主要来源[2] 。
漆酶是一种很有应用潜力的多酚氧化酶,其酶活性在食用菌中最高,一般含有4个铜原子,分布在3个高度保守的不同结合位点,每个铜原子在催化机制中都有很重要的作用。漆酶能催化O2通过4个电子还原成水,并且伴随着一些酚类底物的氧化[13] 。漆酶一般含有500个左右的氨基酸残基,且都有用来指导其有效分泌的信号肽。同一来源的漆酶相似性较高,而不同来源的漆酶其氨基酸序列的相似性并不高,但漆酶在铜离子结合的组氨酸周围都有严格保守的氨基酸残基[15] 。通过对100多个漆酶的序列比对和分析,发现有4个特征性序列区可以用来鉴别漆酶,分别为L1-L4(L1:H–W–H–G–X9–D–G–X5–QCPI; L2: G–T–X–W–Y–H–S–H–X3–Q–Y–C–X–D–G–L–X–G–X–(FLIM); L3:H–P–X–H–L–H–G–H; L4: G–(PA)–W– X–(LFV)–HCHI–DAE–X–H–X3–G–(LMF)–X3–(LFM)[16] )。其中L2和L4符合多铜氧化酶(MCO)含铜蛋白特征性序列,而L1和L3则是漆酶特有的序列区,其中最为保守的漆酶氨基酸序列为L3。通过BlastP比对,发现在漆酶氨基酸序列中发现3个铜氧化酶(Cu-oxidase)保守结构域,分别为Cu-oxidase-3、Cu-oxidase和Cu-oxidase-2。将漆酶氨基酸序列与4种漆酶特征性氨基酸序列进行比对和分析,发现该漆酶基因含有最为保守的漆酶氨基酸序列L3,此序列可以将漆酶与其他的多铜氧化酶区分开,更加证实了该基因片段为漆酶基因。与此同时,应用DNAMAN软件对漆酶氨基酸序列与多种伞菌目白腐菌漆酶氨基酸序列建立了系统进化树,从进化树图中可发现,漆酶氨基酸序列与其他真菌漆酶的同源性最高达到99%,说明漆酶的一级结构序列具有很高的保守性,同为侧耳科的漆酶在亲缘关系上更接近。Piscitelli等[17] 研究认为,Cu2+能诱导漆酶及漆酶基因的转录,是有效的漆酶活性诱导剂,对于铜离子是否参与调节基因的表达,将会在后续试验中进一步研究,本文旨在通过简并PCR技术克隆白灵菇漆酶基因,相对于传统的漆酶基因克隆方法而言[18] -[20] ,简并PCR技术无疑要简捷得多,并对Lacc1基因序列及氨基酸序列进行分析,为进一步研究漆酶酶学性质及该基因的生物学功能和工程菌的构建奠定下理论基础,将为充分开发和利用我国丰富的真菌漆酶贡献一份力量。
致 谢
感谢国家自然科学基金和内蒙古自治区自然科学基金在试验资金方面的资助,感谢郭九峰研究员和那日教授在研究思路和试验指导中的无私帮助和悉心指导。
基金项目
国家自然科学基金(No. 51267014)、内蒙古自治区自然科学基金(No. 2012MS0109)。
参考文献 (References)
- [1] 张金霞, 左雪梅, 黄晨阳 (2003) 食用菌新秀–阿魏侧耳. 土壤肥料, 6, 46-47.
- [2] 朱海潇 (2008) 食用菌产漆酶能力的比较及漆酶性能的研究. 硕士论文, 福建农林大学, 福州.
- [3] 周光龙, 余大廷 (1992) 漆酶与食用菌生产. 中国生漆, 3, 27-30.
- [4] Reinhammar, B. (1984) In: Lontie, R., Ed., Copper Proteins and Cooper Enzymes, Vol. II, CRC Press, Boca Raton.
- [5] Mayer, A.M. (1987) Polyphenol oxidases in plants—Recent progress. Phytochemistry, 26, 11-20.
- [6] Hatakka. (1997) Lignin-modifying enzymes from selected white-rot fungi: Production and role in lignin degradation. FEMS Microbiology Reviews, 13, 125-135.
- [7] Vasilchenko, L.G., Karapetyan, N. and Yachkova, S. (2004) Laccase and cellobiose dehydrogenase. 40, 44-49.
- [8] Valderrama, B., Oliver, P., Medrano-Soto, A., et al. (2003) Evolutionary and structural diversity of fungal laccases. Antonie Van Leeuwenhoek, 84, 289-299.
- [9] Zhang, F.N. and Yu, J. (2000) Decolourisation of acid violet 7 with complex pellets of white rot fungus and activated carbon. Bioprocess Engineering, 23, 295-301.
- [10] Hett, P., Markku, S. and Liisa, V. (2003) Purification, characterization and sequence analysis of laccase from the Ascomycete Mauginiella sp. Enzyme and Microbial Technology, 33, 854-862.
- [11] Galhaup, C. and Haltrich, D. (2001) Enhanced formation of laccase activity by the white-rot fungus Trametet pubescens in the presence of copper. Applied Microbiology and Biotechnology, 56, 225-232.
- [12] 钞亚鹏, 钱世钧 (2001) 真菌漆酶及其应用. 生物工程进展, 5:23-28.
- [13] 胡平平, 付时雨 (2001) 漆酶催化活性中心结构及其特性研究进展. 林产化学与工业, 3, 69-75.
- [14] 董银行, 郭家选 (2012) 葡萄果实β-葡萄糖苷酶基因克隆、原核表达及活性检测. 园艺学报, 6, 1073-1080.
- [15] 张银波, 姜琼, 江木兰, 等 (2004) 金针菇漆酶基因的克隆及其在毕赤酵母中的表达研究. 微生物学报, 6, 775- 779.
- [16] Kumar, S.V., Phale, P.S., Durani, S., et al. (2003) Combined sequence and structure analysis of the fungal laccase family. Biotechnology and Bioengineering, 83, 386-394.
- [17] Piscitelli, A., Giardina, P. and Lettera, V. (2011) Induction and transcriptional regulation of laccases in fungi. Current Genomics, 12, 104-112.
- [18] Kim, S., Leem, Y., Kim, K., et al. (2001) Cloning of an acidic laccase gene (clac2) from Coprinus congregatus and its expression by external pH. FEMS Microbiology Letters, 2, 151-156.
- [19] Perry, C.R., Smith, M., Britnell, C.H., et al. (1993) Identification of two laccase genes in the cultivated mushroom Agaricus bisporus. Journal of General Microbiology, 139, 1209-1218.
- [20] Gouka, R.J., Heiden, M.V., Swarthoff, T., et al. (2001) Cloning of aphenol oxidase gene from Acremonium murorum and its expression in Aspergillus awamori. Applied and Environmental Microbiology, 67, 2610-2616.
附录
白灵菇漆酶基因编码区和非编码区序列及推测的氨基酸序列
Coding and non-coding laccase sequences and speculated amino acids from Pleurotus nebrodensis ATGTTTCCAGGCGCACGGATTCTCGCTACGCTTACATTAGCTCTTCACCTTTTACATGGCACTCATGCTGCCATC 75 M F P G A R I L A T L T L A L H L L H G T H A A I 25 GGGCCCATTGCCGACATGTACATCGTCAACGAGGACGTCTCTCCTGACGGCTTCGCTCGTTCGtgagtggatcct 150 G P I A D M Y I V N E D V S P D G F A R S 46 cggtgtgcttttggccacagcacaaatttattacacaggGCTGTCGTCGCTCGCTCTGTGCTGGCCACAGATCCG 225 A V V A R S V L A T D P 58 ACGCCTGCGTCGGTATCGGTTCCTGGCGTCCTCGTGCAAGGAAACAAGgtaacccatgttcgccttcatgaagcc 300 T P A S V S V P G V L V Q G N K 74 gctcgttttgctcactcttcttagGGCGATAACTTTCAGCTGAACGTTCGCAATCAACTGTCGGACTCGACTATG 375 G D N F Q L N V R N Q L S D S T M 91 TTGAAGACTACTAGTATCgtatgcatgtgaatggttgttttggatgaatactgacttcctgcgcagCGTTGGCAT 450 L K T T S I R W H 100 GGCTTCTTTCAATCCGGCTCTACGTGGGCAGATggtatatcttcactacgtattacgatgcctcgctaatcttct 525 G F F Q S G S T W A D 110 tcaaGGACCCGCGTTCGTGAATCAGTGCCCCATCGCCCCGGGGAATAGCTTCCTgtgagtgtccttaccttgtct 600 G P A F V N Q C P I A P G N S F L 128 ctatctttcattcattgaacattctcagATATGACTTTAACGTTCCCGACCAAGCTGGCACGTTCTGtaagtcga 675 Y D F N V P D Q A G T F W 141 cgatcatgaatccactttgtgcttcctgatcgtatatgcaggGTACCATTCGCATCTTTCCACCCAGTATTGTGA 750 Y H S H L S T Q Y C D 152 TGGTCTTAGAGGACCATTCGTAGTgtaagtttcatatcgataagacgctaagacgcgccgggcttaatcatccac 825 G L R G P F V V 160 gcagATACGATCCCTCCGATCCCCACCTGTCCTTATATGACGTTGACAACGgtgagctgtgaacgtattccggtg 900 Y D P S D P H L S L Y D V D N 175 tctgcaacatgctgacagtctcacttccagCCGACACTGTCATTACACTTGAAGATTGGgtacatttcactccct 975 A D T V I T L E D W 185 ctccctcatggaaattgatgtgttcttactctttctgtagTACCACGTTGCGGCCCCTCAGGGTGCAGTGCTTCC 1050 Y H V A A P Q G A V L P 197 TACTCCTGATAGCACGCTCATTAATGGTAAAGGTCGCTTCGCCGGGGGGCCGACTTCCCCTTTGGCTATCATTAA 1125 T P D S T L I N G K G R F A G G P T S P L A I I N 222 CGTCGAAAGCAACAAGCGATATCGTTTCCGACTTATCTCGATGTCTTGCGACCCCAATTTCACGTTCTCAATCGA 1200 V E S N K R Y R F R L I S M S C D P N F T F S I D 247 CGGTCACTCTTTGCAGGTCATTGAAGCAGATGCTGTCAATATTGTGCCCATCGTCGgtttgtctttcacgctcgg 1275 G H S L Q V I E A D A V N I V P I V 265 cctcactgtcccgattcgctgactatagatggtcgatattgtagTGGATAGTATTCAAATCTTCGCGGGtaagta 1350 V D S I Q I F A G 274 atacccaccctttcgtcaaatctttactaagccgagtttcaaggCCAACGCTATTCATTCGTCCTGAATGCCAAT 1425 Q R Y S F V L N A N 284 CAGGCTGTCGACAATTATTGGATTCGCGCAAATCCCAACTTGGGATCGACTGGtatggcattttgaaagcaacac 1500 Q A V D N Y W I R A N P N L G S T G 302 ttgtgcttcgctgacttcccgtaatgccaggCTTCGAAGGTGGCATNAATTCCGCTATCCTTCGGTATGCTGGTG 1575 F E G G I N S A I L R Y A G 316 CCACTGAAAATGACCCTGCCACGACTTCGTCGACGAGCACTCCCCTTCTGGAGACTAATCTTGTGCCGCTCGAAA 1650 A T E N D P A T T S S T S T P L L E T N L V P L E 341 ATCCTGGTGCTCCTGGTCCAGCTGTCCCTGGAGGCGCAGACATCAACATCAATCTTGCTATGGGCTTCGACTTTA 1725 N P G A P G P A V P G G A D I N I N L A M G F D F 366 CTAACTTTGAAATGACCATCAACGGTacgcagtttaggtcttttcaatgccctggatggctgctcatctacccat 1800 T N F E M T I N G 375 gcccaggtTCCCCCTTCAAAGCACCAACTGgtaagcccaactcaccagcgaaataccaaaaaattgatgtgatat 1875 S P F K A P T 382 tcctgtagCTCCTGTTCTACTCCAGATTCTGTCAGGCGCTACGCCTGCCGCTTCGCTTCTTCCTTCAGGTAGTAT 1950 A P V L L Q I L S G A T P A A S L L P S G S I 405 ATACGCGCTAGAAGCGAACAAAGTTGTCGAAATCTCCATACCCGCCTTAGCTGTCGGAGGACCGgtaagcccaaa 2025 Y A L E A N K V V E I S I P A L A V G G P 426 gcccagcgtcagaaagcatgtgctgacaactatcccgcctgaacagCATCCTTTCCATCTTCACGGAgtgagtaa 2100 H P F H L H G 433 tgcgacacgcacaattttctccagcggctgatcgacctcgtatagCACACGTTCGACGTCATCAGGAGTGCGGGC 2175 H T F D V I R S A G 443 TCTACTACGTATAACTTCGACACCCCTGTGCGACGCGATGTTGTCAACACTGGAATTGACGCGAACGACAACGTT 2250 S T T Y N F D T P V R R D V V N T G I D A N D N V 468 ACCATCCGCTTTGTGACGGATAATCCGGGCCCATGGTTCCTCCACTGgtaggcatttcccgcaatacgtgcgaca 2325 T I R F V T D N P G P W F L H C 484 gccatagactgacagcttcccctagCCACATTGACTGGCATCTCGAAATgtaggtggcattctttattgattcaa 2400 H I D W H L E I 492 ttactcgactcaaaggcatttagCGGTCTCGCGGTCGTTTTCGCCGAAGATGTGGCGTCCATCAAGGCCCCGCC 2474 G L A V V F A E D V A S I K A P P 509 TggtatgcctcttcgtatctatccaccgcagcctgtgcgtatatgctgaccttcatttctccagCCGCGTGGGAC 2549 A A W D 513 GACTTGTGTCCGATTTATGATGCTTTGAGCGATTCCGACAGAGGTGGCATAGCTTAG 2606

NOTES
*通讯作者。
